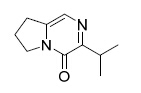
 图 1
Provalin (1)结构图
Figure 1.
Structure of provalin (1)
图 1
Provalin (1)结构图
Figure 1.
Structure of provalin (1)

Citation: Kang Wen-Jia, Wu Sheng, Hua Huiming, Pan Hai-Xue, Tang Gong-Li. Discovery of a New Pyrazinone Natural Product by Genome Mining[J]. Chinese Journal of Organic Chemistry, 2016, 36(7): 1696-1699. doi: 10.6023/cjoc201602016

基于基因组挖掘的一个新的吡嗪酮类天然产物的发现
English
Discovery of a New Pyrazinone Natural Product by Genome Mining
-
Key words:
- pyrazinone
- / Streptomyces
- / genome mining
- / biosynthesis
-
吡嗪酮(pyrazinone)类天然产物是由微生物产生的含非芳香吡嗪酮环的化合物, 目前已发现的这类天然产物包括:耐甲氧西林金黄色葡萄球菌(MRSA)产生的tyrvalin, phevalin和leuvalin[1, 2], 链霉菌产生的phevalin, arglecin, argvalin, maremycin E/F, JBIR-56/-57[3~6], 粘细菌产生的nannozinone A/B和sorazinone B[7], 疣孢菌产生的butrepyrazinone[8], 以及类芽孢杆菌产生的paenibacillin A[9].吡嗪酮类天然产物的抗菌活性较弱, 研究发现其作为信号分子在MRSA等病原菌的感染中起着关键作用[1, 10]; 同时, 一些经化学衍生的吡嗪酮类化合物是微管蛋白、丙型肝炎NS3蛋白酶等靶标的良好抑制剂[11, 12, 13].因此, 发现具有新颖骨架结构的吡嗪酮化合物具有重要意义.
随着基因测序技术的进步和生物合成研究的深入, 近年来出现了一种全新的天然产物发现模式, 即基于生物信息指导发现新化合物的方法——基因组挖掘.已有研究表明, 吡嗪酮天然产物的核心骨架由非核糖体肽合成酶(nonribosomal peptide synthetases, NRPSs)催化形成, 两分子氨基酸通过双模块NRPS活化和缩合后, 在末端还原酶结构域(reductase, RE)的作用下还原解离并环化[2, 14].本课题组采用基因组扫描的方法寻找可能负责新吡嗪酮天然产物生物合成的基因, 通过基因操作、对比发酵、化合物分离和结构鉴定, 从链霉菌Streptomyces sp. TP-A0365中发现一个新的吡嗪酮类天然产物provalin (图 1).与之前发现的此类化合物的氨基酸组成不同, provalin由脯氨酸和缬氨酸缩合并环化形成5~6并环结构.本研究结果为该类化合物的化学衍生提供了新的选择, 同时也展示了基因组挖掘在定向发现新天然产物方面的高效性.
1 结果与讨论
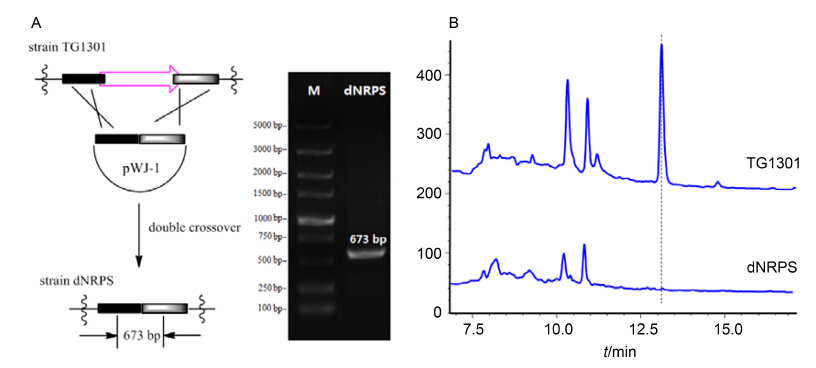
本课题组在分析链霉菌Streptomyces sp. TP-A0365的基因组时发现, 其中包含一个两模块NPRS的编码基因, 其结构域组成与已知吡嗪酮类天然产物的NPRS相似, 且蛋白同源性较高, 但是NRPS识别的两个氨基酸底物与已知的该类化合物不同, 推测其编码一个新的吡嗪酮类天然产物.由于该链霉菌是天然产物yatakemycin的产生菌, 为了排除此类化合物的干扰, 我们选择敲除了yatakemycin生物合成基因簇的菌株TG1301作为原始菌株[15], 采用同源重组双交换的方法敲除NRPS基因, 经聚合酶链式反应(PCR)验证获得基因型正确的突变株命名为dNRPS(图 2A).采用原始发酵条件[15], 将突变株dNRPS与TG1301同时发酵.发酵粗提液的高效液相色谱(HPLC)分析结果显示, 与TG1301相比, dNRPS突变株不再产生保留时间13.1 min的化合物1 (图 2B). LC-MS分析显示该化合物m/z 179.1 [M+H]+与推测的脯氨酸和缬氨酸底物缩合形成的的吡嗪酮天然产物的分子量相符.该化合物同时也存在于野生型链霉菌Streptomyces sp. TP-A0365的发酵产物中.
将菌株TG1301进行大量发酵(20 L), 并通过反向硅胶及半制备高效液相色谱分离纯化获得了化合物1.该化合物为无色固体, 紫外光谱于230, 320 nm有强吸收, 高分辨质谱(HR-ESIMS)给出其准分子离子峰m/z 179.1177 [M+H]+, 提示其分子式为C10H14N2O (计算值179.1184 [M+H]+).
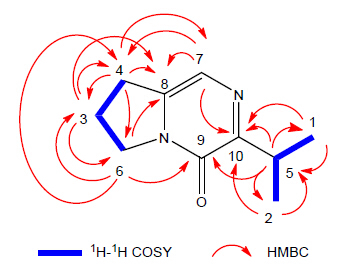
化合物1的1H NMR谱(溶剂是DMSO-d6)存在两个甲基氢信号(δH 1.10, 1.12), 三个亚甲基氢信号(δH 2.10, 3.04, 3.98), 一个脂肪链次甲基氢信号(δH 3.28), 一个烯烃氢信号(δH 7.25). 13C NMR谱显示有10个碳信号:结合DEPT谱能指认其中两个甲基碳信号(δH 20.2, 20.2, 重叠); 三个仲碳信号(δH 21.0, 28.8, 48.5);两个叔碳信号, 其中一个为烯烃碳(δH 117.2), 另一个为脂肪链次甲基碳(δH 29.3);三个季碳(δH 141.4, 154.3, 158.6)(表 1).通过HSQC谱, 我们能确定相关的碳信号对应的氢信号, 1H-1H COSY谱中, H-5分别与H-1, H-2; H-3分别与H-4、H-6有氢氢相关. HMBC谱中, H-1, H-2, H-5都与C-10相关, 由此能确定这一部分连接在C-10位.另外H-3与C-4, C-6, C-8; H-4与C-3, C-6, C-7, C-8; H-6与C-3, C-4, C-8, C-9都有相关信号, 由此能确定这部分吡咯环结构并且得出氮原子连接着羰基.烯烃的氢原子H-7与C-4, C-8, C-10也有远程相关, 更加确证了这一结构(图 3).根据以上信息, 化合物1的结构鉴定为: 3-异丙基-7, 8-二氢吡咯并[1, 2-a]吡嗪-4(6H)-酮, 命名为provalin (图 1).
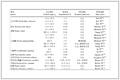 表 1
化合物1核磁共振图谱数据(500/125 MHz, DMSO-d6)
Table 1.
NMR spectroscopic data of compound 1 (500/125 MHz, DMSO-d6)
表 1
化合物1核磁共振图谱数据(500/125 MHz, DMSO-d6)
Table 1.
NMR spectroscopic data of compound 1 (500/125 MHz, DMSO-d6)
Position δC δH HMBC (H→C) 1H-1H COSY 1 20.2 q 1.10 (s, 3H) C-5, 10 H-5 2 20.2 q (重叠) 1.12 (s, 3H) C-5, 10 H-5 3 21.0 t 2.10 (m, J=7.5 Hz, 2H) C-4, 6, 8 H-4, 6 4 28.8 t 3.04 (t, J=7.5 Hz, 2H) C-3, 6, 7, 8 H-3 5 29.3 d 3.28 (m, J=7.0 Hz, 1H) C-1, 2, 9, 10 H-1, 2 6 48.5 t 3.98 (t, J=7.5 Hz, 2H) C-3, 4, 8, 9 H-3 7 117.2 d 7.25 (s, 1H) C-4, 8, 10 8 141.4 s 9 154.3 s 10 158.6 s 表 1 化合物1核磁共振图谱数据(500/125 MHz, DMSO-d6)
Table 1. NMR spectroscopic data of compound 1 (500/125 MHz, DMSO-d6)2 实验部分
2.1 仪器与试剂、生物材料
PCR扩增仪Eppendorf Mastercycler, 凝胶成像系统BioRad Syngene, 质谱仪Thermo Scientific LCQ Fleet Ion Trap, 高效液相色谱仪Agilent 1260, 高分辨质谱仪Agilent Technologies 6230 TOF LC/MS, 核磁共振仪Bruker AVANCE DRX-500, 分析色谱柱Diamonsil C18 5 μ 250 mm×4.6 mm, 半制备柱Venusil XBP C18, 5 μm, 100 Å, 250 mm×10 mm.生物试剂购自Takara公司, 引物合成和DNA测序由苏州金唯智公司完成, 进口化学试剂购自Sigma-Aldrich公司, 国产化学试剂购自上海国药.链霉菌Streptomyces sp. TP-A0365由日本富山县立大学的Yasuhiro Igarashi教授提供, 菌株TG1301由本实验室构建, 其它大肠杆菌和质粒由本实验室保存.
2.2 构建NRPS基因敲除突变株
在pKC1139载体中构建含左右臂的同框敲除质粒pWJ-1, 左臂引物为PYR-L1 (GAATTCCAACTGCTGC-TCGGCACCTG)和PYR-L2 (TCTAGAGCCGTAGCC-GAGTCGAACGTC), 右臂引物为PYR-R1 (TCTAGAGACGTACCTGCCGGAAGAGTG)和PYR-R2 (AAGCTTCTGACCGAGTTCTTCACCAC).同框敲除质粒构建、接合转移及突变株筛选方法参考文献[15], 通过PCR验证突变株的基因型, 验证引物为PYR-V1 (CACTCACAGGAAGGCAGGA)和PYR-V2 (TCGTTGCGCAGATGTCCGGAG), 目标条带大小为673 bp, 获得正确的NRPS基因同框敲除突变株dNRPS.
2.3 突变株的发酵和化合物1的提取和分离
菌株TG1301和dNRPS采用文献中的方法发酵[15], 收集50 mL发酵液菌体用3倍体积丙酮浸泡并超声10 min, 减压浓缩至干, 用0.5 mL DMSO溶解, 样品离心去沉淀后供HPLC和LC-MS分析.收集20 L菌株TG1301的发酵液菌体, 用丙酮提取2次, 离心去沉淀, 上清旋转蒸发除去丙酮, 剩余水相用乙酸乙酯萃取2次, 有机相减压浓缩得浸膏, 用DMSO溶解浸膏, 经反向硅胶柱初步分离纯化, 半制备高效液相色谱进一步纯化[V(水):V(乙腈)=7:3~2:8, 流速3 mL/min, 检测波长320 nm, 最终得化合物1 (15 mg).
2.4 发酵结果分析与化合物1结构鉴定
HPLC分析:将菌株dNRPS与TG1301的发酵粗提液分别用HPLC和LC-MS检测, 通过对比出峰情况确定NRPS基因敲除后菌株发酵产物的变化, 以此确定TG1301发酵产物中的目标化合物. HPLC条件: A相为水(含0.1%甲酸), B相为乙腈(含0.1%甲酸), 流速1 mL/min, 检测波长320 nm及全波长扫描, 洗脱梯度0~3 min, 10% B; 3~10 min, 10%~40% B; 10~21 min, 40%~80% B; 21~25 min, 80%~10% B. LC-MS采用与HPLC相同的条件.对于纯化得到的化合物1, 进行高分辨质谱和NMR分析, 得到化合物完整的结构信息.
辅助材料(Supporting Information)化合物1的紫外吸收光谱, MS, HR-ESIMS, NMR图谱.这些材料可以免费从本刊网站(http://sioc-journal.cn/)上下载.
-
-
[1]
Wyatt, M. A.; Wang, W.; Roux, C. M.; Beasley, F. C.; Heinrichs, D. E.; Dunman, P. M.; Magarvey, N. A. Science 2010, 329, 294. doi: 10.1126/science.1188888
-
[2]
Zimmermann, M.; Fischbach, M. A. Chem. Biol. 2010, 17, 925. doi: 10.1016/j.chembiol.2010.08.006
-
[3]
Alvarez, M. E.; White, C. B.; Gregory, J.; Kydd, G. C.; Harris, A.; Sun, H. H.; Gillum, A. M.; Cooper, R. J. Antibiot. 1995, 48, 1165. doi: 10.7164/antibiotics.48.1165
-
[4]
Tatsuta, K.; Fujimoto, K.; Yamashita, M.; Tsuchiya, T.; Umezawa, S. J. Antibiot. 1973, 26, 606. doi: 10.7164/antibiotics.26.606
-
[5]
Tang, Y.-Q.; Sattler, I.; Thiericke, R.; Grabley, S.; Feng, X.-Z. Eur. J. Org. Chem. 2000, 2, 261.
-
[6]
Motohashi, K.; Inaba, K.; Fuse, S.; Doi, T.; Izumikawa, M.; Khan, S. T.; Takagi, M.; Takahashi, T.; Shinya, K. J. Nat. Prod. 2011, 74, 1630. doi: 10.1021/np200386c
-
[7]
Jansen, R.; Sood, S.; Mohr, K. I.; Kunze, B.; Irschik, H.; Stadler, M.; Müller, R. J. Nat. Prod. 2014, 77, 2545. doi: 10.1021/np500632c
-
[8]
Kyeremeh, K.; Acquah, K. S.; Camas, M.; Tabudravu, J.; Houssen, W.; Deng, H.; Jaspars, M. Mar. Drugs 2014, 12, 5197.
-
[9]
Bian, X.; Shao, M.; Pan, H.; Wang, K.; Huang, S.; Wu, X.; Xue, C.; Hua, H.; Pei, Y.; Bai, J. Nat. Prod. Res. 2016, 30, 125. doi: 10.1080/14786419.2015.1041941
-
[10]
Secor, P. R.; Jennings, L. K.; James, G. A.; Kirker, K. R.; Pulcini, E. D.; McInnerney, K.; Gerlach, R.; Livinghouse, T.; Hilmer, J. K.; Bothner, B.; Fleckman, P.; Olerud, J. E.; Stewart, P. S. PLoS One 2012, 7, e40973.
-
[11]
Taggi, A. E.; Stevenson, T. M.; Bereznak, J. F.; Sharpe, P. L.; Gutteridge, S.; Forman, R.; Bisaha, J. J.; Cordova, D.; Crompton, M.; Geist, L.; Kovacs, P.; Marshall, E.; Sheth, R.; Stavis, C.; Tseng, C. P. Bioorg. Med. Chem. 2016, 24, 435. doi: 10.1016/j.bmc.2015.08.038
-
[12]
Gising, J.; Belfrage, A. K.; Alogheli, H.; Ehrenberg, A.; Åkerblom, E.; Svensson, R.; Artursson, P.; Karlén, A.; Danielson, U. H.; Larhed, M.; Sandström, A. J. Med. Chem. 2014, 57, 1790. doi: 10.1021/jm301887f
-
[13]
Zhang, X.; Glunz, P. W.; Jiang, W.; Schmitt, A.; Newman, M.; Barbera, F. A.; Bozarth, J. M.; Rendina, A. R.; Wei, A.; Wen, X.; Rossi, K. A.; Luettgen, J. M.; Wong, P. C.; Knabb, R. M.; Wexler, R. R.; Scott Priestley, E. Bioorg. Med. Chem. Lett. 2013, 23, 1604. doi: 10.1016/j.bmcl.2013.01.094
-
[14]
Wyatt, M. A.; Mok, M. C.; Junop, M.; Magarvey, N. A. Chembiochem 2012, 13, 2408. doi: 10.1002/cbic.v13.16
-
[15]
Huang, W.; Xu, H.; Li, Y.; Zhang, F.; Chen, X.-Y.; He, Q.-L.; Igarashi, Y.; Tang, G.-L. J. Am. Chem. Soc. 2012, 134, 8831.
-
[1]
-
表 1 化合物1核磁共振图谱数据(500/125 MHz, DMSO-d6)
Table 1. NMR spectroscopic data of compound 1 (500/125 MHz, DMSO-d6)
Position δC δH HMBC (H→C) 1H-1H COSY 1 20.2 q 1.10 (s, 3H) C-5, 10 H-5 2 20.2 q (重叠) 1.12 (s, 3H) C-5, 10 H-5 3 21.0 t 2.10 (m, J=7.5 Hz, 2H) C-4, 6, 8 H-4, 6 4 28.8 t 3.04 (t, J=7.5 Hz, 2H) C-3, 6, 7, 8 H-3 5 29.3 d 3.28 (m, J=7.0 Hz, 1H) C-1, 2, 9, 10 H-1, 2 6 48.5 t 3.98 (t, J=7.5 Hz, 2H) C-3, 4, 8, 9 H-3 7 117.2 d 7.25 (s, 1H) C-4, 8, 10 8 141.4 s 9 154.3 s 10 158.6 s -

 扫一扫看文章
扫一扫看文章
计量
- PDF下载量: 0
- 文章访问数: 2906
- HTML全文浏览量: 520


 下载:
下载:


 下载:
下载:

