 图1
LFOP示意图
Figure1.
Schematic diagram of a LFOP instrument
图1
LFOP示意图
Figure1.
Schematic diagram of a LFOP instrument

Citation: Zhang Aiqin, Wang Man, Zhang Hui, Jin Jun, Shen Gangyi. Advances in Label-Free Optical Biosensor[J]. Chemistry, 2018, 81(1): 21-28, 44.

免标记光学生物传感器研究进展
English
Advances in Label-Free Optical Biosensor
-
生物传感器具有灵敏度高、选择性好、特异性强、分析速度快、动态在线分析等特点,在食品分析、环境监测、药物研发、生化检测、医学诊断等领域中得到了广泛应用[1~3]。作为生物传感器中的一类,免标记型光学生物传感器(Label-free optical biosensor,LFOP)以光单元为信号转换元件,以生物探针分子为识别传感元件,将待测物与探针分子发生特异性反应所产生的生物信号实时转换为光信号,从而实现定性定量检测,其原理示意见图 1。LFOP无需对分析物进行标记或衍生,既保持了被测分子原有性质和状态,又避免了复杂的标记过程及其造成的假阳信号等不利影响;而且分析过程样品的活性可有效保持,不会产生化学或物理破坏,便于多次分析。鉴于此,它越来越受到科研工作者广泛关注与重视[4~6]。
当前关于LFOP的报道虽呈蜂拥之势,遗憾的是相关系统性总结不多。鉴于此,本文对LFOP的发展状况进行总结,并就它们的性能和发展前景进行论述,以期为该领域的进一步发展提供参考。
1 免标记型光学生物传感器
LFOP类型丰富多样,但应用于实际检测的并不多。究其根源,除光学原理、仪器构造等限制因素外,方法技术的特色性以及应用领域的宽广性等至关重要。基于此,本文重点选择了当前发展成熟且具代表性的四种LFOP,即基于全反射倏逝波[7, 8]与波导层膜发生耦合作用原理的表面等离子体共振(Surface plasmon resonance,SPR)传感器、光波导光模光谱(Optical waveguide lightmode spectroscopy,OWLS)传感器、椭圆偏振光学(Spectroscopic ellipsometry,SE)传感器以及基于膜干涉原理[9, 10]的反射干涉光学(Reflectometry interference spectroscopy,RIFS)传感器展开介绍和论述。
1.1 SPR生物传感器
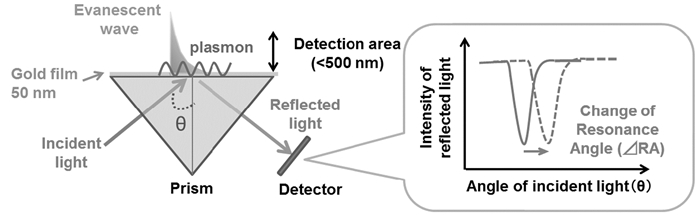
它是基于SPR理论而建立的光分析技术,Liedberg等[11]首次将之用于生化检测。SPR的核心原理[12~14]如图 2所示,一束p-偏振光以大于临界角的入射角度从光密介质(玻璃棱镜)射向光疏介质(分析物)形成全内反射。入射光部分深入到光疏介质形成倏逝波。在光密介质和光疏介质界面加一层厚约50nm的金膜。满足一定入射角度时,倏逝波将和金膜波导层的表面等离子体发生共振,导致入射光能量被吸收,而反射光强度瞬时衰减。基于金膜上分析物折射率不同发生共振所引起角度不同的原理,进行定性定量分析。
1990年,Pharmacia公司首次推出商品化Biacore系列仪器。截止目前已有多家仪器公司生产出了成熟的SPR检测仪,代表性的有Biacore系列、XPR 36仪等。
从方法学角度看,SPR传感器目前主要向提高检测灵敏度、扩大分析通量、发展新方法如局域SPR(LSPR)、SPR成像(SPRi)及仪器微型化、联用化等方向发展[15~18]。Xue等[17]把氧化石墨烯修饰于SPR芯片的金膜表面,利用若丹明6G(R6G)分子可诱导石墨烯光学性能变化的原理实现了R6G超灵敏检测,检测限(LOD)低至10-17mol/L。SPRi可解决SPR不能进行高通量分析的瓶颈[19]。
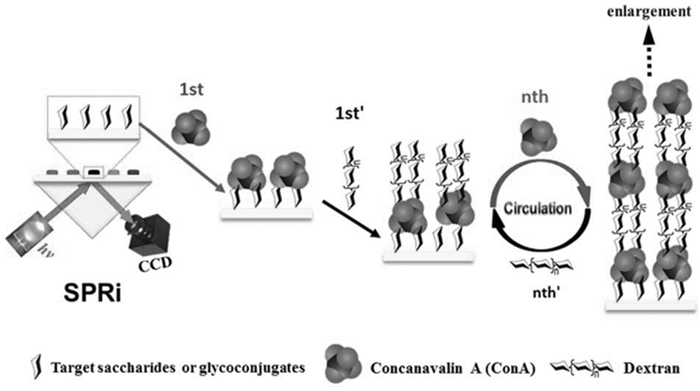
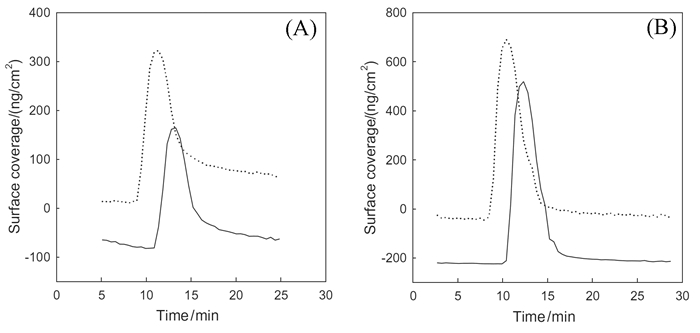
陈义课题组[20~22]致力于高灵敏SPRi仪器的开发,他们先后发展了小分子糖的SPRi高灵敏检测方法,如图 3所示。利用刀豆球蛋白A和葡聚糖对糖分子的循环识别和多步扩增,可使葡萄糖的LOD低至2.5μmol/L,其灵敏度甚至高出SPR法[21]。最近,他们又实现了亚飞摩尔级含量Micro-RNA的高灵敏检测。对于血清中的肿瘤标志物Micro-RNA-15a,其检测灵敏度比常规SPRi提高了约107倍。相关研究对于如何提高LFOP的灵敏度具有参考意义。
SPR已广泛用于生物分析、药物研发、食品安全与环境监测、医学临床诊断等众多领域。在生物分析方面,基因组学和蛋白质组学是SPR研究及应用的重点方向。Katz等[23]分别采用荧光光谱法、SPR和酶联免疫吸附(ELISA)法考察了ASPP2Ank-SH3与Bcl-2的结合,结果显示SPR法的灵敏度明显高于荧光光谱法。王洋等[24]采用化学沉积法在L-半胱氨酸修饰过的金膜表面合成了针对血红蛋白的分子印迹聚合物膜,利用SPR对血红蛋白进行检测的LOD可低至0.05mg/mL。杨帆等[25]采用SPR研究了肿瘤坏死因子(TNF)与其胞外两种可溶性受体(TNF sRI/Fc Chimera和TNF sRII/Fc Chimera)的相互作用,结果显示它们的亲和力远大于常规的抗原-抗体的作用力。Gamsjaeger等[26]采用SPR竞争法,结合稳态模式,定量分析了蛋白和DNA的相互作用。Song等[27]以信号转导和转录激活因子3(STAT3)蛋白为靶标,利用LSPR建立了抗肺癌药物的筛选模型。笔者课题组[28, 29]也开展了生命体大分子蛋白对氨基酸手性识别研究。如图 4所示,利用SPR技术可动态直观地监测蛋白对小分子氨基酸对映异构体的高效识别[28],对于探索和揭示生命体内生物分子对氨基酸的手性选择和识别具有一定意义。
在食品安全与环境监测方面,SPR传感器可用于病原体、毒素、药物残留、化学污染物等的检测。Melaine等[30]在金膜表面构筑DNA探针,利用SPRi并结合纳米金信号扩增高灵敏检测了绿脓杆菌、沙门氏嗜肺军团菌的特征核苷酸序列16S rRNA,其线性范围为0.01~100 ng/mL。Song等[31]利用LSPR检测了食品中的大肠杆菌O157:H7,基于核壳型纳米金-SiO2结构的信号放大效应,使得O157:H7的LOD低至10CFU。Xia等[32]利用SPR开展了牛奶等成分复杂食品中抗菌素的检测。采用免疫抑制法,能检测出实际样品中浓度为5.28、2.26 ng/mL的氯霉素和庆大霉素。
在医学临床诊断方面,Li等[33]把微流控和SPR结合起来构建了微通道光纤SPR传感器,进行了人血中葡萄糖的微量快速监测。前列腺特异性抗原(PSA)是前列腺癌的重要标志物,Denizli等[34]把PSA的单克隆抗体固定在芯片金膜上,利用SPR对临床样品中的PSA进行了检测。其LOD低至18×10-14mol/L,且方法选择性很高。Ribaut等[35]则通过SPR检测癌症相关生物信号小分子来进行肺癌诊断。
1.2 OWLS生物传感器
相对于SPR以贵金属金作为信号转换的波导层,OWLS常采用SiO2-TiO2等金属氧化物高折射率材料。此外OWLS还采用光栅衍射耦合模式来克服平面薄膜双折射导致灵敏度降低的现象。OWLS的原理如图 5所示[36],光源发出的光经光栅衍射耦合进入波导层,在其内部发生全反射;波导层表面固定的生物敏感膜的不同,会引起倏逝波与波导层内传输的光波强度、相位和频率的变化。测量这些量的变化可以获得生物膜的信息。
1995年,Lukosz首次把OWLS用于生化检测[37]。目前已有Microvacuum等公司推出了商品化仪器。OWLS的发展方向集中于提高检测灵敏度、仪器微型化、联用化等[38~40]。例如,Kehl等[39]将OWLS与角度可调制的微反射镜系统结合,有效扩展了光扫描范围,提高了检测灵敏度。OWLS的研究领域主要集中在生物分析、食品安全与环境监测、生物膜等方面。
在生物分析方面,吴中伟等[41, 42]利用OWLS研究了固液界面表皮生长因子与其受体作用的动力学行为以及DNA-DNA结合蛋白的相互作用。Orgovan等[43]动态监测了人类血液分化出的原代单核细胞的吸附和铺展行为。Madsen等[44]在研究牛下颚粘蛋白的降解和该过程对其在疏水表面的吸附、润滑作用的影响时,用OWLS准确揭示了其吸附特性。Vert等[45, 46]用OWLS监测了生理环境中白蛋白、球蛋白等蛋白的粘附行为,发现聚乙二醇化可减少白蛋白的沉降,对于研究改善外源性材料周围的血液相容性具有重要意义。
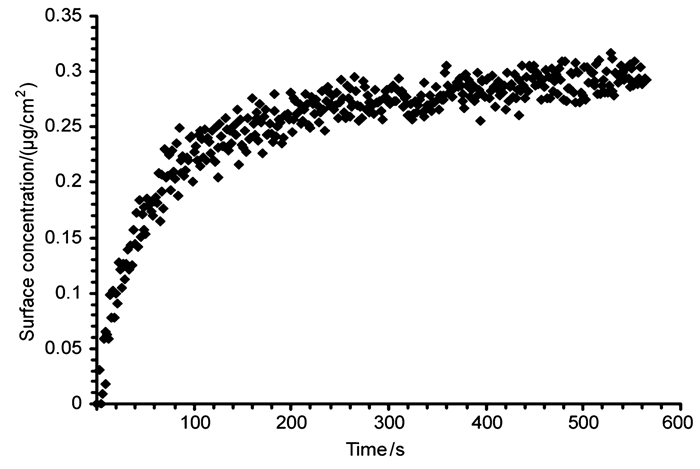
在食品安全与环境监测方面,宋保栋等[47]利用所建立的OWLS开展了饮用水中杀虫剂2, 4-二氯苯氧乙酸(2, 4-D)的检测,可完全满足我国相关检测标准要求。吐尔地等[48]对挥发性有机污染物苯乙烯、二甲苯、甲苯等蒸汽气体进行了检测。利用OWLS能够检测到体积分数仅为1×10-9的苯乙烯蒸汽,且传感器可逆性好。Adányi等[49]在食品检测方面开展了相关研究,鲤鱼中卵黄蛋白原测定的线性范围为600~12000 ng/mL,最低检出范围为3~100 ng/mL。他们还进行了小麦中致癌物脱氧雪腐镰刀菌烯醇的检测[50]。Kroslak等[51]采用OWLS监测了咖啡提取物纳米级小粒子在表面沉积的动态过程。Szalontai等[52]实时监测了发酵乳发酵过程中双歧杆菌O1356和嗜酸乳杆菌O1132两种益生菌含量的动态变化行为。Kim等[53]把多克隆抗多酚氧化酶抗体固定于OWLS传感器芯片表面,开展了食品中多酚氧化酶的测定。如图 6所示,在20min内实现了马铃薯和葡萄中多酚氧化酶的含量检测。该方法LOD可低至0.005607U/mL。
Majer-Baranyi等[54]检测了两栖类物种蟾蜍中外源性雌激素生物标记物卵黄蛋白原的含量。采用直接测定模式,卵黄蛋白原的线性范围为0.1~10 μg/mL;若采用竞争性模式,线性范围可降至0.5~50 ng/mL。
在膜材料领域,Wu等[55]利用OWLS评价了3种阳离子型多肽在聚氧化乙烯层上的有序竞争性吸附行为。Beatrix等[56]动态监测了正负电荷纳米金粒子层层组装(LBL)的过程,对于深入理解LBL的作用机制具有一定意义。陆祖宏等[57]利用OWLS动态监测了银纳米颗粒晶种在巯丙基三甲氧基硅烷修饰的SiO2-TiO2波导层表面的吸附和生长过程,对于研究膜表面银纳米颗粒的生长控制因素具有重要意义。
1.3 SE生物传感器
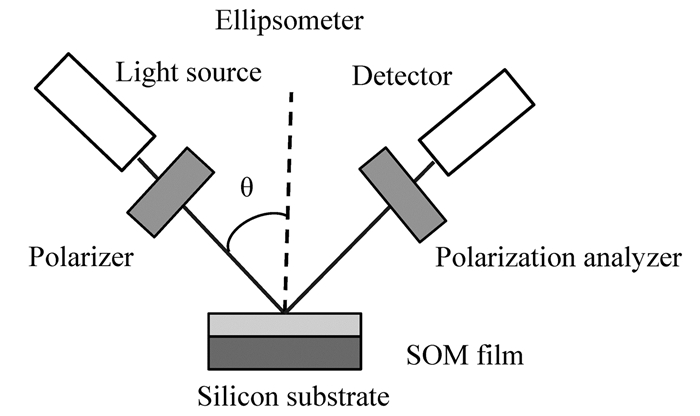
SE是在椭圆偏振光学原理发展起来的一种生物传感技术。基本原理如图 7所示[58],一束线偏振光入射至硅基底薄膜表面,全内反射下膜表面产生倏逝波。传感膜在对待测生物分子吸附或特异性结合的同时对倏逝波进行调制,导致反射光偏振态改变(由线偏振态变为椭圆偏振态)。偏振态改变与生物分子的膜厚、折射率等相关,基于此变化可获得生物分子信息。
早在1945年,Rothen[59]就已首次将SE技术用于膜性能测定,目前已有JA Wallam等公司推出商品化仪器。有关SE技术的研究集中于提高检测灵敏度、扩大检测通量以及发展新方法如SE成像、红外SE等[60~62],主要应用于生物分析、材料科学等领域。
在生物分析方面,SE应用于和生物医学相关如抗原-抗体、配体-受体相互作用,蛋白类激素、病毒以及肿瘤标记蛋白检测。Mustafa等[63]利用SE研究了β-淀粉样肽(Aβ1-16)与单克隆DE2抗体的免疫反应,发现它们有强特异性作用,KD可达1.46×10-8mol/L。Lu等[64]把鼠抗人PSA单克隆抗体固定于波导层SiO2的表面,利用SE研究了它与PSA的作用。靳刚课题组[65~67]利用SE及SE成像在生物医学检测等领域做了大量工作。他们研究了两种中和单克隆抗体h12和b1与血清中SARS冠状病毒(SARS-CoV)的相互作用,其中,SARS-CoV和h12的作用过程如图 8所示,其KA(1.36×107L/mol)大于后者(9.5×106L/mol)。他们还发现h12抗体在非还原性条件下与SARS-S1蛋白结合,而在还原性条件下不结合,因此h12能识别寡聚S1蛋白表面的抗原决定簇。相关研究为中和抗体的生产和SARS疫苗的研制提供了依据。
糖抗原(CAl5-3)是监测乳腺癌的重要标志性物质,Zhang等[68]搜集了60名乳腺癌患者的血清,利用SE对血清中CAl5-3进行了定量检测,结果与电化学发光免疫法有很好的一致性。Cakmak等[69]则利用SE实时研究了血浆蛋白在双模两亲性网络聚合物β-APCNs上的动态吸附行为。
由于SE中偏振态变化与膜的厚度和折射率有关,相比前两种LFOP,其能够获取更多的膜性能信息。Gobsch等[70]利用SE监测了有机太阳能电池活性层膜中高度有序的各层聚合物的形态及分布。Leick等[71]原位监测了基于原子层沉积法制备铂系金属超薄膜的制备过程,既可准确测出不同元素的原子成膜厚度,还可精确判断出它们在沉积过程中不同的成核延迟点。还有学者[72~75]利用SE研究了各种手性聚合物膜、金属氧化膜、无机膜等的性质。此外,SE还可应用于其他光学传感器的传感芯片中波导材料的性能表征与测定。
传感金膜的性能对SPR传感器的灵敏度等至关重要。Al-Rubaye等[76]在进行LSPR生物传感器研究时,利用SE研究了金膜的光学性能,为LSPR开展小分子检测所需纳米结构材料的金膜的制备提供了依据。
1.4 RIFS生物传感器
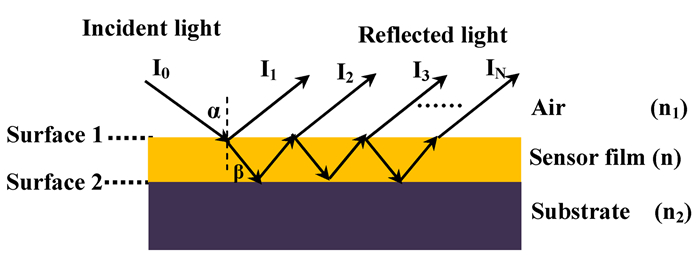
RIFS传感器是20世纪90年代初由Gauglitz等[77]首次建立的一种生物传感技术,其主要基于光学薄膜干涉原理发展而来。原理如图 9所示,一束光以小于全反射角射到平行平面薄膜上,在膜两表面上发生多次反射和折射。当满足一定条件时可简化该模型,即入射光线I0一部分在界面1上发生反射产生I1;另一部分透过界面1折射,并在界面2上反射,然后该反射光线又在界面1上射出,形成I2。I1与I2产生光程差而形成干涉。如改变薄膜厚度,反射干涉光谱就会在极大值和极小值之间变化。根据极值的位置(波长大小)即可测定膜厚。在基片上固定某种生物分子配体,当有相应分析物与之结合时会实时引起膜厚度的改变。该厚度与浓度变化线性相关,据此进行定量分析。
RIFS目前多集中于提高检测灵敏度的研究。传统的薄膜干涉传感单元主要由玻璃基底和用于构筑生物分子的表面干涉薄膜层组成。常规干涉层材料的折射率偏低,致使检测灵敏度较低。李建林[78]在基底和干涉层间构筑了一层厚度可控的干涉增强层TiO2,可明显改善系统的信噪比和提高其灵敏度。Segal等[79, 80]则把干涉层硅片的表面刻蚀成多孔型硅纳米结构,其大的比表面积和多样化的修饰功能为生物配体的固定提供了良好环境。以其作为传感单元所制备的生物传感器显示了优良的稳定性和快速的再生性,在复杂生物体液以及活细胞检测中优势明显。
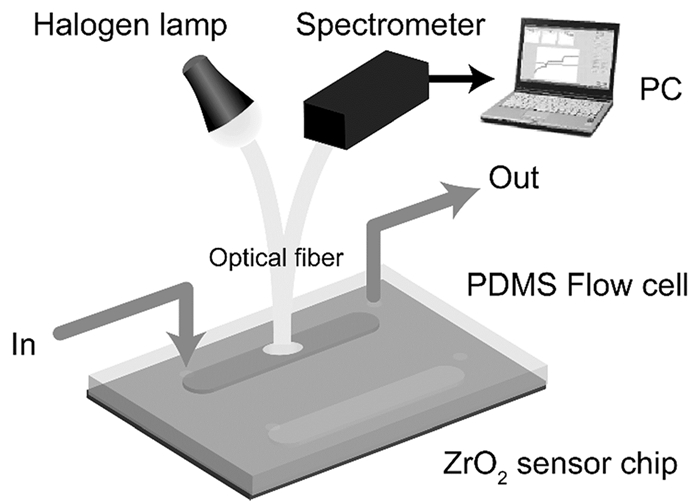
RIFS生物传感器在生物分析、食品安全与环境监测及材料科学等领域应用较常见。在生物分析方面,Takeuchi等[81~84]在蛋白质相互作用领域做了大量工作。他们结合微流控技术,研制了微流控-RIFS传感器(图 10)。以ZrO2为干涉膜传感单元,研究了多药耐药性膜蛋白MRP1与其抗体的相互作用[81]。他们[82]还以SiN为传感单元干涉层,结合光化学通过多层修饰在其表面固定C-反应蛋白抗体,检测C-反应蛋白质(CRP)时的LOD可低至100ng/mL,完全满足血清中CRP的检测要求。Li等[85]则基于人造细胞膜,利用RIFS研究了蛋白离子通道的功能。
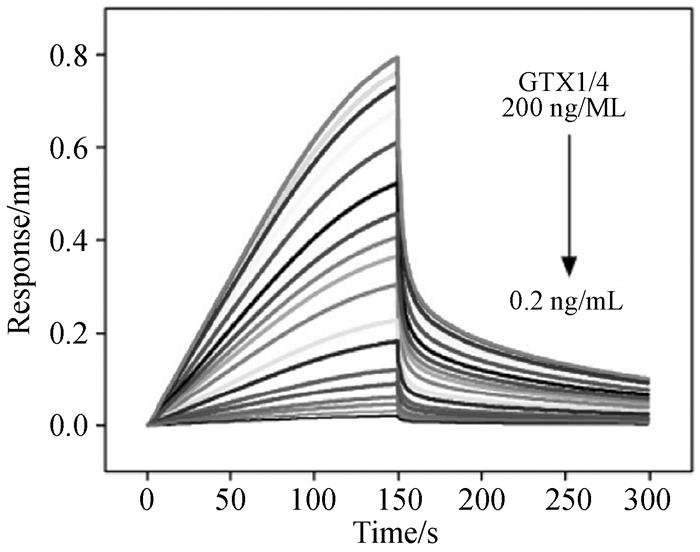
在食品安全与环境监测方面,膝沟藻毒素(gonyautoxin)1/4(GTX1/4)作为全球海洋生物三大危害之一麻痹性贝毒的典型,给人类的健康和生命带来严重威胁。Wang等[86]利用氧化石墨烯-配体指数富集系统进化筛选技术(GO-SELEX)筛选出了对GTX1/4具有高度特异性亲和的小分子核酸适体GO18-T-d,将其构筑于干涉膜上制成生物传感器,用于GTX1/4检测。结果显示该传感器不仅具有高度选择性,而且还有宽的检测范围(0.2~200 ng/mL,图 11)和低的LOD(50pg/mL)。该传感器可实际用于了海洋生物尖刺贝类中GTX1/4的检测。
Tang等[87, 88]应用FT-RIFS开展了食品中疾病菌及抗生素的快速检测。Rau等[89]利用RIFS首次开展了成分复杂且低浓度的牛奶样品中睾酮激素的检测。对于实际样品,其LOD为94.9ng/L,测量过程仅耗时15min,完全满足实际检测需要。Sebok等[90]基于LBL法,经过20次重复循环,在玻璃基底表面构筑了ZnO2/聚苯乙烯磺酸钠(PSS)纳米杂化膜(厚约514nm)。将其作为干涉层用于乙醇蒸汽的检测,可显著改善信噪比,LOD降低了5倍。结合丁基三氯硅烷(修饰于杂化膜表层),可使灵敏度提高10倍,而LOD降低3倍。
在材料科学方面,廖荣等[91]结合薄膜分析软件SCOUT,利用RIFS进行了多种固体薄膜厚度的测量。Huang等[92]研究了氟碳化合物封端的聚碳酸酯型聚氨酯和聚苯乙烯两种材料表面蛋白质的竞争吸附行为。
2 性能比较
上述四种LFOP技术的使用流程基本相同。通常先将某种分子作为配体或者探针固定于传感单元的芯片表面,再将待检样品溶液与其接触发生作用,通过作用前后引起光信息的变化实现对样品检测。
同时,由于光学原理差异,它们各有优点和不足。SPR适用范围广,除了能测分子相互作用的动力学常数之外还可测得热力学常数以及物质浓度,是当前研究蛋白质等生物大分子相互作用最常用的工具之一,也是四种LFOP技术中研究和应用最多的一种。但其只能得到样品质量信号的变化信息,而无法获取其结构信息;并且以贵金属金为主的基底材料制作要求高,成本贵。OWLS采用竞争性免疫抑制反应时灵敏度为ELISA的1000倍,且本底噪声小,适合在线原位分析;但系统相对复杂,光源构造要求高,稳定性相对差。SE能精确测出尺寸厚度为Å的原子层膜,获得分子质量、膜厚度等多种信息,并且波导层基底材料要求不高。不足之处在于只能获得样品的相对厚度而非绝对值,且系统构造相对复杂。RIFS系统构造相对简单,易实现微型化;但其灵敏度不高,误差较大,多用于静态分析,在动态监测方面有待进一步开发。
3 展望
作为集光学、生物、化学与医学等为一体的传感技术,LFOP独特的优点为深入推进生命科学研究提供了新平台。其研究和应用范围非常广,几乎延伸到与生命相关的所有领域,是目前最为活跃的研究领域之一。
然而,LFOP也存在一些不足。最近及将来一段时间内,除了针对各自不足而展开个性化研究外,还有些共性问题需深入探究:(1)进一步提高和改善LFOP灵敏度。相比其他类型传感器,LFOP检测灵敏度还有待提升,这也是其当前的一个主要研究点。针对此,重点在以传感单元为核心,开发新的高性能波导层纳米材料、发展新型的生物功能探针固定化技术和信号放大技术等。例如,陈义课题组发展的循环放大技术非常有借鉴意义;(2)LFOP之间不同光学技术的联用。目前已有一些基于两种光学原理结合而发展的新技术,如平面光波导干涉技术(Integrated planar optical waveguide interferometer)、表面等离子体共振椭圆偏振技术(SPR Ellipsometry,SPRE)以及双偏振干涉(Dual-polarization interferometry,DPI)等[93~96]。DPI把倏逝波和干涉两种光学技术优势结合,能精确测量样品的厚度、密度和质量绝对值,大大拓展了其功能。杨秀荣课题组[97~99]在DPI生物检测做了大量创新性研究;(3)发展与其他分析检测技术的联用,弥补其性能不足。例如,SPR-MS、SPR-Raman以及SE-AFM等联用技术[100~102];(4)生物检测范围及对象的深入和扩大,特别是开展原位和活体检测(In situ and in vivo detection);(5)基于光学原理的不可穷尽特性,发展新LFOP方法和技术;(6)仪器微型化、集成化、智能化研究;(7)商品化仪器的研发和推广。
可以相信LFOP将会有更广阔的应用和发展前景,它不但能推动传统传感技术的发展,而且将对临床诊断、医药学研究、食品卫生和环境监测、免疫学、材料学等诸多科学研究领域都会产生深远影响。
-
-
[1]
S K Arya, C C Wong, Y J Jeon et al. Chem. Rev., 2015, 115(11):5116~5158. doi: 10.1021/cr500554n
-
[2]
X Wang, J Hao. Sci. Bull., 2016, 61(16):1281~1295. doi: 10.1007/s11434-016-1151-6
-
[3]
J Kirsch, C Siltanen, Q Zhou et al. Chem. Soc. Rev., 2013, 42:8733~8768 doi: 10.1039/c3cs60141b
-
[4]
X Wang, O S Wolfbeis. Anal. Chem., 2016, 88:203~227. doi: 10.1021/acs.analchem.5b04298
-
[5]
O Bleher, A Schindler, M X Yin et al. Anal. Bioanal. Chem., 2014, 406:3305~3314. doi: 10.1007/s00216-013-7504-y
-
[6]
J K Chen, G Y Zhou, C J Chang et al. Sens. Actuat. B, 2014, 194:10~18. doi: 10.1016/j.snb.2013.12.071
-
[7]
F Chiavaioli, P Biswas, C Trono et al. Anal. Chem., 2015, 87:12024~12031. doi: 10.1021/acs.analchem.5b01841
-
[8]
M Brändén, S Dahlin, F Höök. ChemPhysChem, 2008, 9(11):2480~2485.
-
[9]
M Citartan, S C B Gopinath, J Tominaga et al. Analyst, 2013, 138:3576~3592. doi: 10.1039/c3an36828a
-
[10]
K S Mun, S D Alvarez, W Y Choi et al. ACS Nano, 2010, 4(4):2070~2076. doi: 10.1021/nn901312f
-
[11]
B Liedberg, C Nylander, I Lunström. Sensor. Actuat., 1983, 4:299~304. doi: 10.1016/0250-6874(83)85036-7
-
[12]
J Homola. Chem. Rev., 2008, 108(2):462~493. doi: 10.1021/cr068107d
-
[13]
杨雨曦, 孙雅婷, 杨梦朝等.生命科学仪器, 2014, 12:7~16. http://www.cqvip.com/qk/86265x/201405/662905183.html
-
[14]
Y Yanase, T Hiragun, K Ishiiet al. Sensors, 2014, 14:4948~4959. doi: 10.3390/s140304948
-
[15]
王霄, 许吉英, 陈义.化学进展, 2015, 27(5):550~558. http://kns.cnki.net/KCMS/detail/detail.aspx?filename=hxjz201505010&dbname=CJFD&dbcode=CJFQ
-
[16]
P Singh. Sensor. Actuat. B, 2016, 229:110~130. doi: 10.1016/j.snb.2016.01.118
-
[17]
T Xue, S Yu, X Zhang et al. Sci. Rep., 2016, 6(2):77~80.
-
[18]
A R Ferhan, J A Jackman, N J Cho. Anal. Chem., 2017, 89(7):4301~4308. doi: 10.1021/acs.analchem.7b00370
-
[19]
申刚义, 陈义, 张轶鸣等.化学进展, 2010, 22:1648~1655. http://www.cqvip.com/QK/71135X/201107/34831370.html
-
[20]
J Chen, J Y Xu, Y Chen. Chin. Chem. Lett., 2013, 24:651~653. doi: 10.1016/j.cclet.2013.04.006
-
[21]
C J Liu, X Wang, J Y Xu et al. Anal. Chem., 2016, 88(20):10011~10018. doi: 10.1021/acs.analchem.6b02184
-
[22]
F Hu, J Xu, Y Chen. Anal. Chem., 2017, 89(18):10071~10077. doi: 10.1021/acs.analchem.7b02838
-
[23]
C Katz, H Benyamini, S Rotem et al. PNAS, 2008, 105(34):12277~12282. doi: 10.1073/pnas.0711269105
-
[24]
王洋, 任亚敏, 张庆文等.科学通报, 2014, 59(9):779~786. http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=gdxxhxyj201401010
-
[25]
杨帆, 杨秀荣.分析化学, 2013, 41(5):664~669. http://www.cqvip.com/QK/93919X/201305/47538733.html
-
[26]
R Gamsjaeger, R Kariawasam, L H Bang et al. Anal. Biochem., 2013, 440:178~185. doi: 10.1016/j.ab.2013.04.030
-
[27]
S Song, A H Nguyen, J U Lee et al. Analyst, 2016, 141:2493~2501. doi: 10.1039/C5AN02397A
-
[28]
申刚义, 高妍, 崔箭等.高等学校化学学报, 2011, 32(8):1709~1713. http://kns.cnki.net/KCMS/detail/detail.aspx?filename=gdxh201108011&dbname=CJFD&dbcode=CJFQ
-
[29]
申刚义, 高妍, 崔箭等.高等学校化学学报, 2015, 36(5):881~885. http://kns.cnki.net/KCMS/detail/detail.aspx?filename=gdxh201505011&dbname=CJFD&dbcode=CJFQ
-
[30]
F Melaine, M Saad, S Faucher et al. Anal. Chem., 2017, 89:7802~7807. doi: 10.1021/acs.analchem.7b01942
-
[31]
L P Song, L Zhang, Y J Huang et al. Sci. Rep., 2017, 7:3288. doi: 10.1038/s41598-017-03495-1
-
[32]
Y Q Xia, R X Su, R L Huang et al. Biosens. Bioelectron., 2016, 92:266~272.
-
[33]
D C Li, B Y Lu, R Zhu et al. Biomicrofluidics, 2016, 10(1):011913. doi: 10.1063/1.4942946
-
[34]
G Ertürk, H Özen, M A Tümer et al. Sens. Actuat. B, 2016, 224:823~832. doi: 10.1016/j.snb.2015.10.093
-
[35]
C Ribaut, V Voisin, V Malachovská et al. Biosens. Bioelectron., 2016, 77:315~322. doi: 10.1016/j.bios.2015.09.019
-
[36]
R Konradi, M Textor, E Reimhult. Biosensors, 2012, 2:341~376. doi: 10.3390/bios2040341
-
[37]
W Lukosz. Sens. Actuat. B, 1995, 29(1-3):37~50. doi: 10.1016/0925-4005(95)01661-9
-
[38]
谢骁, 刘全俊, 陆祖宏.激光与光电子学进展, 2006, 43(11):33~42. http://kns.cnki.net/KCMS/detail/detail.aspx?filename=jgdj200611008&dbname=CJFD&dbcode=CJFQ
-
[39]
F Kehl, G Etlinger, T E Gartmann et al. Sens. Actuat. B, 2016, 226:135~143. doi: 10.1016/j.snb.2015.11.072
-
[40]
X W Zhang, L Liu, L Xu. Appl. Phys. Lett., 2014, 104(3):033703. doi: 10.1063/1.4861596
-
[41]
吴中伟, 刘全俊, 吴凌伟等.光谱学与光谱分析, 2010, 30(10):2776~2779. doi: 10.3964/j.issn.1000-0593(2010)10-2776-04
-
[42]
吴中伟, 刘全俊, 吴凌伟等.光学学报, 2007, 27(3):521~525. http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=gxxb200703027
-
[43]
N Orgovan, R Salánki, N Sándor et al. Biosens. Bioelectron., 2014(54):339~344. http://www.sciencedirect.com/science/article/pii/S0956566313007823
-
[44]
J B Madsen, B Svensson, M Abou Hachem et al. Langmuir, 2015, 31(30):8303~8309. doi: 10.1021/acs.langmuir.5b01281
-
[45]
M Vert. Eur. Polym. J., 2015, 68:516~525. doi: 10.1016/j.eurpolymj.2015.03.051
-
[46]
L Leclercq, E Modena, M J Vert. Biomater Sci. Polym. Ed., 2013, 24:1499~518. doi: 10.1080/09205063.2013.772045
-
[47]
宋保栋, 施汉昌, 何苗等.中国环境科学, 2007, 27(3):360~364. http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=zghjkx200703015
-
[48]
姑力米然·吐尔地, 艾尔肯·吐尔逊, 阿曼古丽·图尔贡等.应用化学, 2015, 32(2):232~238. doi: 10.11944/j.issn.1000-0518.2015.02.140161
-
[49]
N Adányi, K Majer-Baranyi, A Nagy et al. Sens. Actuat. B, 2013, 176:932~939. doi: 10.1016/j.snb.2012.10.079
-
[50]
K Majer-Baranyi, A Székács, I Szendrö et al. Eur. Food Res. Technol., 2011 233:1041~1047. doi: 10.1007/s00217-011-1598-2
-
[51]
M Kroslak, D Morbidelli. Chem. Pap., 2014, 68(12):1755~1766.
-
[52]
H Szalontai, A Nóra, A Kiss. New Biotechnol., 2015, 31:395~401.
-
[53]
N Kim, W-Y Kim. Food Chem., 2015, 169:211~217. doi: 10.1016/j.foodchem.2014.07.130
-
[54]
K Majer-Baranyi, N Adányi, A Nagy et al. Environ. Anal. Chem., 2015, 95:481~493. doi: 10.1080/03067319.2015.1048434
-
[55]
X Wu, P R Matthew, M Joseph et al. Colloid. Surf. B, 2015, 130:69~76. doi: 10.1016/j.colsurfb.2015.04.011
-
[56]
P Beatrix, K Sandor, P Daniel. J. Am. Chem. Soc., 2014, 30:13478~13482.
-
[57]
Z W Wu, Q J Liu, L W Wu et al. Adv. Sci. Lett., 2011, 4:516~521. doi: 10.1166/asl.2011.1217
-
[58]
P Liu, Y Zhang, S T Martin. Environ. Sci. Technol., 2013, 47:13594~13601. doi: 10.1021/es403411e
-
[59]
A Rothen. Rev. Sci. Instrum., 1945, 16(2):26~30. doi: 10.1063/1.1770315
-
[60]
S Engmann, V Turkovic, H Hoppe et al. J. Phys. Chem., 2013, 117(47):25205~25210.
-
[61]
H Gu, X Chen, H Jiang et al. J. Opt., 2016, 18:025702. doi: 10.1088/2040-8978/18/2/025702
-
[62]
A Furchner, G Sun, H Ketelsen et al. Analyst, 2015, 140:1791~1797 doi: 10.1039/C4AN01853B
-
[63]
M K Mustafa, A Nabok, D Parkinson et al. Biosens. Bioelectron., 2013, 26(4):1332~1336.
-
[64]
X Zhao, F Pan, B Cowsill et al. Langmuir, 2011, 27(12):7654~7662. doi: 10.1021/la201245q
-
[65]
C Qi, J Z Duan, Z H Wang et al. Biomed. Microdevices, 2006, 8(3):247~253. doi: 10.1007/s10544-006-8305-2
-
[66]
Y Niu, J Zhuang, X Yan et al. Thin Solid Films, 2011, 59(9):2768~2771.
-
[67]
C Qi, Y Lin, J Feng et al. Virus Res., 2009, 140:79~84. doi: 10.1016/j.virusres.2008.11.010
-
[68]
H G Zhang, C Qi, Z Wang et al. Clin. Chem., 2005, 51(6):1038~1040. doi: 10.1373/clinchem.2004.043240
-
[69]
G Guzman, S M Bhaway, T Nugay et al. Langmuir, 2017, 33(11):2900~2910. doi: 10.1021/acs.langmuir.7b00281
-
[70]
S Engmann, V Turkovic, H Hoppe et al. J. Phys. Chem., 2013, 117(47):25205~25210.
-
[71]
N Leick, J W Weber, A J M Mackus et al J. Phys. D:Appl. Phys., 2016, 49:115504. doi: 10.1088/0022-3727/49/11/115504
-
[72]
G J Lee, E H Choi, W K Ham et al. Opt. Mater. Express, 2016, 6(3):767~781. doi: 10.1364/OME.6.000767
-
[73]
H He, D A Andersson, D D Allred et al J. Phys. Chem. C, 2013, 117:16540~16551. doi: 10.1021/jp401149m
-
[74]
K S Tseng, Y L Lo. Opt. Mater. Express, 2014, 4(1):43~56. doi: 10.1364/OME.4.000043
-
[75]
P Liu, Y Zhang. Environ. Sci. Technol., 2013, 47:13594~13601. doi: 10.1021/es403411e
-
[76]
A G Al-Rubaye, A Nabok, A Tsargorodska. Sens. Bio-Sens. Res., 2017, 12:30~35. doi: 10.1016/j.sbsr.2016.11.006
-
[77]
G Gauglitz, W Nahm. J Fresenius. Anal. Chem., 1991, 341(3-4):279~283. doi: 10.1007/BF00321563
-
[78]
李建林.分析化学, 2008, 36(6):831~834.
-
[79]
K Urmann, J-G Walter, E Segal. Anal. Chem., 2015, 87:1999~2016. doi: 10.1021/ac504487g
-
[80]
K Urmann, S Arshavsky-Graham, J G Walter et al. Analyst, 2016, 141:5432~5440. doi: 10.1039/C6AN00810K
-
[81]
Y Kurihara, T Sawazumi, T Takeuchi. Analyst, 2014, 139:6016~6021. doi: 10.1039/C4AN00925H
-
[82]
H W Choi, Y Sakata, Y Kurihara et al. Anal. Chim. Acta, 2012, 728:64~68. doi: 10.1016/j.aca.2012.03.030
-
[83]
Y Kurihara, M Takama, M Masubuchi et al. Biosens. Bioelectron., 2013, 40:247~251. doi: 10.1016/j.bios.2012.07.032
-
[84]
Y Kurihara, M Takama, T Sekiya et al. Langmuir, 2012, 28:13609~13615. doi: 10.1021/la302221y
-
[85]
Z Li, Y Tang, L Zhang et al. Lab Chip, 2014, 14:333~341. doi: 10.1039/C3LC50937K
-
[86]
S Gao, B Hu, X Zheng et al. Biosens. Bioelectron., 2016, 79:938~944. doi: 10.1016/j.bios.2016.01.032
-
[87]
Y Tang, Z Li, Q Luo et al. Biosens. Bioelectron., 2016, 79:715~720. doi: 10.1016/j.bios.2015.12.109
-
[88]
Y Tang, Z Li, J Liu et al. Anal. Chem., 2013, 85:2787~2794. doi: 10.1021/ac303282j
-
[89]
S Rau, G Gauglitz. Anal. Bioanal. Chem., 2012, 402(1):529~536. doi: 10.1007/s00216-011-5470-9
-
[90]
D Sebök, I Dékany. Sens. Actuat. B, 2015, 206:435~442. doi: 10.1016/j.snb.2014.09.087
-
[91]
廖荣, 张海燕, 杨铁铮等.中国测试, 2013, 39(6):10~13.
-
[92]
Y Huang, X Lu, W Qian et al. Acta Biomater., 2010, 6(6):2083~2090. doi: 10.1016/j.actbio.2009.12.035
-
[93]
K Peter, F Kehl, E Ehrentreich-Förster et al. Biosens. Bioelectron., 2014, 58:287~307. doi: 10.1016/j.bios.2014.02.049
-
[94]
A Garcíamarín, J M Abad, E Ruiz et al. Anal. Chem., 2014, 86:4969~4976 doi: 10.1021/ac5005212
-
[95]
C J Cirák, K Bombarová. Mater. Today:Proceed., 2015, 2(1):70~76. doi: 10.1016/j.matpr.2015.04.010
-
[96]
M Trevor, T P Mcnamara, F Fan et al. ACS Appl. Mater. Interf., 2015, 7(45):25270~25280. doi: 10.1021/acsami.5b07290
-
[97]
T Hu, S Wang, C Chen et al. Anal. Chem., 2012, 84:924~930. doi: 10.1021/ac2019443
-
[98]
Y Zheng, Y Cheng, F Yang et al. Anal. Chem., 2014, 86:3849~3855. doi: 10.1021/ac403992r
-
[99]
T Hu, S Wang, C Chen et al. Anal. Chem., 2017, 89:2606~2612. doi: 10.1021/acs.analchem.6b04950
-
[100]
J Breault-Turcot, P Chaurand, J-F Masson. Anal. Chem., 2014, 86:9612~9619. doi: 10.1021/ac502077b
-
[101]
S A Meyer, B Auguié, E C L Ru et al. J. Phys. Chem. A, 2012, 116(3):1000~1007. doi: 10.1021/jp2107507
-
[102]
E Finot, L Markey, F Hane et al. Colloid Surf. B, 2013, 104(1):289~293.
-
[1]
-
-

 扫一扫看文章
扫一扫看文章
计量
- PDF下载量: 0
- 文章访问数: 0
- HTML全文浏览量: 0


 下载:
下载:










 下载:
下载:

